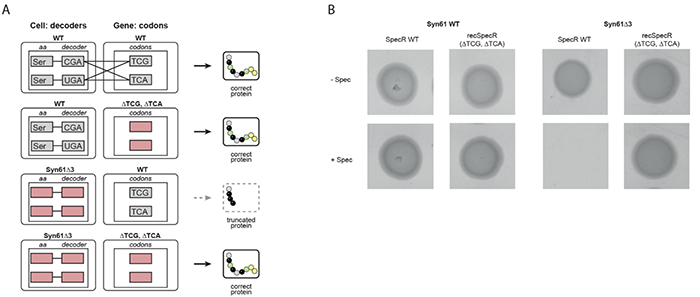
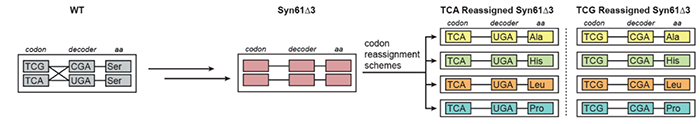
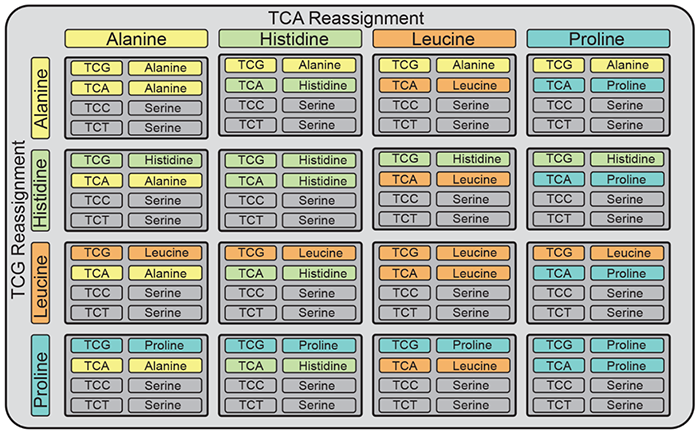
密码子这一通用的遗传密码定义了基因与蛋白质氨基酸之间的对应关系。正是因为所有形式的生命基本上都采用相同的遗传密码,进化创新可以通过生物之间水平基因转移(Horizontal gene transfer,HGT)的方式来共享,这是进化的主要驱动因素。水平基因转移指的是将遗传物质传递给其他细胞而非子代的过程,比如细胞的接合、转导以及转化等等。与之相对应的基因垂直转移,指的是生物将基因遗传给子代的过程。转座子、病毒、质粒等移动遗传因子,利用遗传密码的通用性以及宿主细胞内的装置读取自己的基因,以牺牲宿主细胞的方式进行繁殖。维持共同的遗传密码可以允许病原体等以通过水平基因转移的方式获得有益于生存的条件。线粒体和叶绿体中存在一些对标准遗传密码的偏差,绝大多数的特征密码子涉及到了终止密码子【1-2】。但目前所知的细胞核基因组中密码子重置的还很少。因此,如果能进行密码子的重分配,将有助于创建一个“遗传防火墙”,限制遗传信息的水平转移。为了检测该假设的可能性,英国剑桥大学Jason W. Chin研究组在Science发文题为Refactored genetic codes enable bidirectional genetic isolation,重构了大肠杆菌的遗传密码结构,并创建了正交遗传密码,从而可以限制合成遗传信息逃逸到自然生命体之中。基因组合成和编辑技术给了重写遗传密码的可能性,并可以用于创造具有新解码工具的有机体。作者们首先合成了一个4Mb的大肠杆菌基因组,并定义了一个新的密码子规则。作者们将其中TCG和TCA丝氨酸密码子替换为同义的AGC和AGT密码子,还用TAA替换了TAG终止密码子,从而产生了Syn61菌株【3】。作者们进一步地进化了该菌株,删除了解码TCG以及TCA密码子的tRNAs以及在TAG终止密码子处与终止蛋白质RF-1的合成基因prfA,从而得到了Syn61∆3菌株,几乎不能读取通用遗传密码中的所有密码子。进一步地,作者们用压缩的遗传密码创建了一个重编码大霉素耐药基因recSpecR (∆TCG, TCA),recSpecR (∆TCG, TCA)使得Syn61∆3菌株具有大霉素的抗药性(图1)。压缩的遗传密码子使用的是规范密码子的子集。因此,用规范代码编写的遗传信息可以在解码规范代码的细胞中被读取,但不能在全基因组编码压缩和同源tRNA缺失的细胞中读取。如果能够重新编写遗传信息的解码规范,将能够限制遗传信息从工程生物系统向自然系统的转移。作者们发现野生型F质粒能够有效转移到标准遗传密码读取的细胞中,但是不能转移到Syn61∆3菌株中。将野生型F质粒突变成为包含tRNA基因serT的质粒后,则可以水平基因转移到Syn61∆3菌株之中。说明serT的获得足以绕过Syn61∆3中编码压缩和同源tRNA缺失提供的遗传隔离。基于此,作者们希望进一步重新分配密码子的,在Syn61∆3菌株中中独立地将TCA和TCG密码子重新分配给Ala、His、Leu或Pro,从而创建了16个新的遗传密码子(图2)。在每一个新的遗传密码中,作者们都改变了编码特定意义密码子上的规范氨基酸的身份。该重新分配策略类似于自然进化中为密码子捕获模型。通过重构遗传密码的结构,新遗传密码将用于编码Ala和Pro的密码子数量从4个增加到6个,将用于编码His的密码子数量从2个增加到4个,并将主要用于编码Leu的密码子数量从6个增加到8个。当创建一个合成基因的遗传码解码器使得其与自然基因的规范遗传码解码器对功能正交,可那么合成基因中可以被正交解码器正确读取,但是不能被自然界的规范解码器读取,从而阻止合成基因向自然界的流动。总的来说,几乎通用的遗传密码定义了基因中的密码子和蛋白质中的氨基酸之间的对应关系,而作者们工作的重构了大肠杆菌的遗传密码结构,并创建了正交遗传密码,以限制合成遗传信息逃逸到自然生命。作者们开发了一种正交和相互正交的水平基因转移系统,允许遗传信息在使用相同遗传密码的生物之间转移,但限制遗传信息在使用不同遗传密码的生物之间转移,阻碍移动遗传元素的错误入侵。
原文链接:
https://www.science.org/doi/10.1126/science.add8943
1. M. Kollmar, S. Mühlhausen, Nuclear codon reassignments in the genomics era and mechanisms behind their evolution. BioEssays 39, 1600221 (2017). doi:10.1002/bies.201600221 Medline
2. L. Borges, Y. C. Lou, R. Sachdeva, B. Al-Shayeb, P. I. Penev, A. L. Jaffe, S. Lei, J. M. Santini, J. F. Banfield, Widespread stop-codon recoding in bacteriophages may regulate translation of lytic genes. Nat. Microbiol. 7, 918–927 (2022). doi:10.1038/s41564-022-01128-6 Medline
3. K. Wang, J. Fredens, S. F. Brunner, S. H. Kim, T. Chia, J. W. Chin, Defining synonymous codon compression schemes by genome recoding. Nature 539, 59–64 (2016). doi:10.1038/nature20124 Medline